Stammzellenbasierte Embryomodelle: KI verbessert Selektion und Vorhersagbarkeit
-
Quelle:
IAI, KIT - News 2025
- Datum: 21.02.2025, aktualisiert 12.03.2025
-
Für ausführliche Informationen siehe
KIT - Das KIT - Medien - News - News 2025 - KI erkennt normal entwickelte Embryos
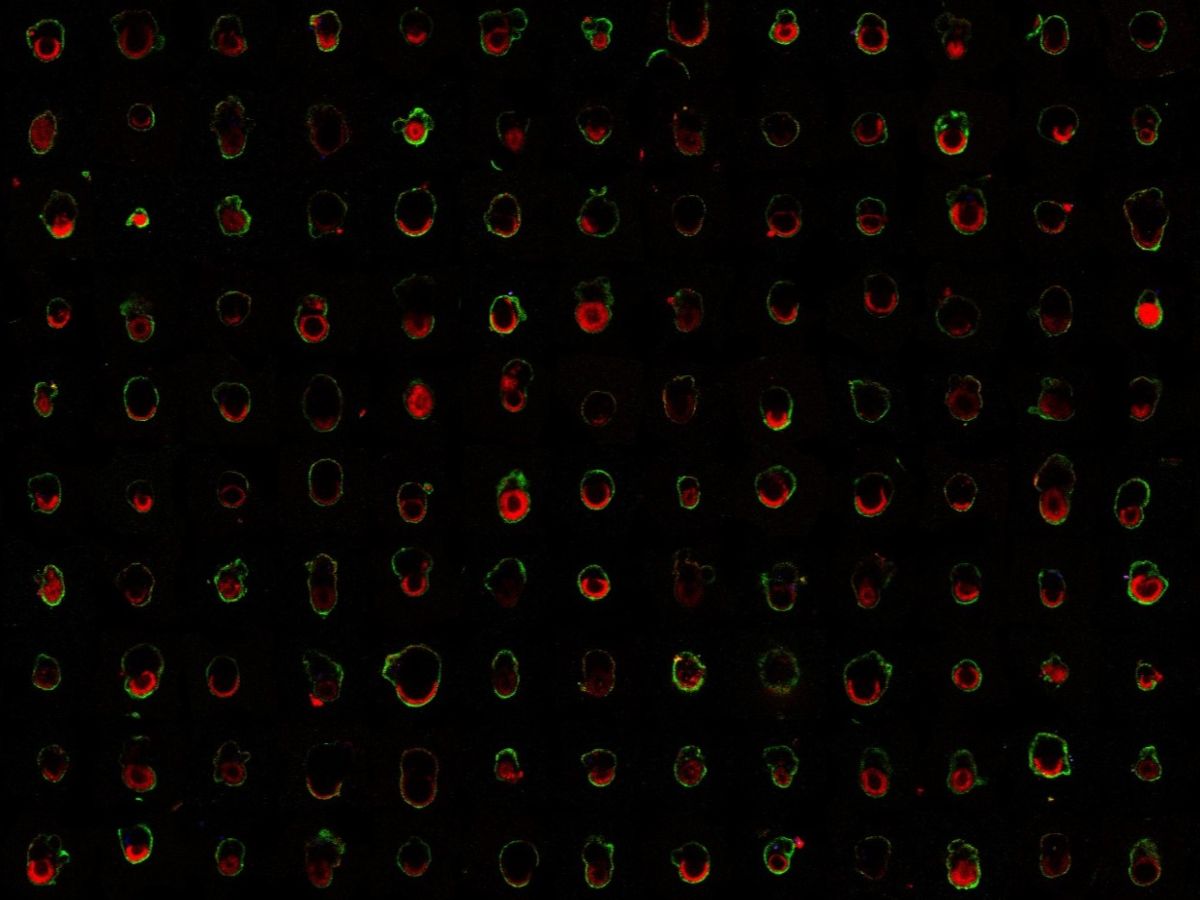
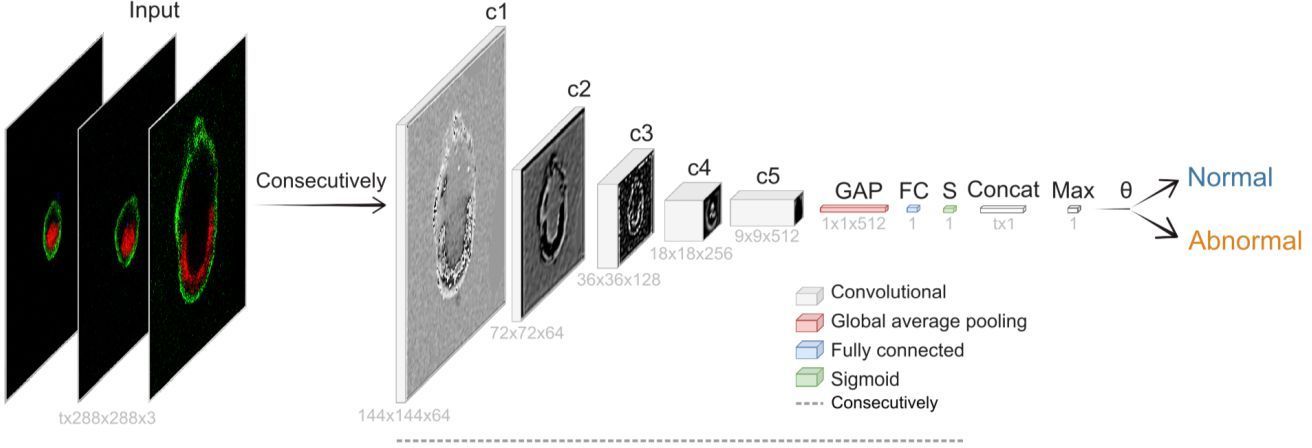
Stammzellenbasierte Embryomodelle sind ein leistungsstarkes Werkzeug zur Erforschung der frühen Entwicklung, doch ihre Variabilität stellt eine Herausforderung für die Standardisierung der Forschung dar. Der Doktorand Luca Deininger und ein Team am Caltech haben Deep Learning eingesetzt, um die Reproduzierbarkeit bei der Auswahl von ETiX-Embryos zu verbessern. Sie klassifizierten 900 Strukturen in normale und abnormale Kategorien. Mithilfe von Live-Imaging und KI-basierten Modellen erreichten sie eine Genauigkeit von 88 % nach 90 Stunden Zellkultur und 65 % bereits zum Zeitpunkt der sog. Zellenseedung, um Entwicklungsverläufe vorherzusagen. Die Studie identifiziert zentrale Merkmale normal entwickelter ETiX-Embryos, darunter höhere Zellzahlen, größeres Volumen und eine kompakte Morphologie, und bestätigt durch Perturbationsexperimente, dass eine Erhöhung der initialen Zellanzahl die Entwicklung normaler Embryos begünstigt.
Diese Arbeit wurde zu je 50 % der Personal- und Reisekosten von der Helmholtz-Gemeinschaft im Rahmen der Graduiertenschule „HIDSS4Health“ (Helmholtz Information and Data Science School for Health) und dem Helmholtz-Programm NACIP (Natural, Artificial and Cognitive Information Processing) gefördert. Das High Performance Computing (HPC) auf der HAICORE@KIT-Partition wurde durch den Impuls- und Vernetzungsfonds der Helmholtz-Gemeinschaft unterstützt.
Das Team veröffentlichte seine Ergebnisse in der Fachzeitschrift Nature Communications (https://doi.org/10.1038/s41467-025-56908-5).

