3D+t Terabyte Bildanalyse
- Ansprechperson:
- Projektgruppe:
- Förderung:
DFG (assoziiertes Projekt zum SPP 1736 Algorithms for Big Data)
- Projektbeteiligte: Prof. Dr. Peter Sanders (KIT, Institut für Theoretische Informatik, Algorithmik II)
- Starttermin:
2015
Beschreibung:
Im Fokus des Projekts steht die Entwicklung von Algorithmen für die Analyse von Zeitreihen hochauflösender 3D-Bilder. Das Ziel sind neue Bildanalysemethoden, die sowohl hohe Qualität als auch Recheneffizienz gewährleisten und die Möglichkeiten moderner Hardware effizient ausnutzen.
Die Leistungsfähigkeit der entwickelten Methoden soll an realen Datensätzen von Zebrabärblingsembryos demonstriert werden, die durch Lichtscheibenmikroskopie beobachtet werden. Solche Datensätze erreichen Größen über 10 Terabyte pro Embryo, was zwei Größenordnungen über der Leistungsfähigkeit bisher vorgeschlagener konkurrierender Algorithmen liegt. Zusätzlich sollen Benchmarkdatensätze mit vergleichbarer Struktur erzeugt und analysiert werden, bei denen die wirklichen Objektbewegungen bekannt sind, um eine quantitative Bewertung der Ergebnisse zu gestatten.
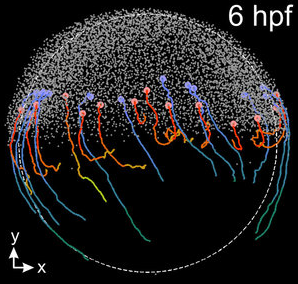
Kobitski, A.; Otte, J. C.; Takamiya, M.; Schäfer, B.; Mertes, J.; Stegmaier, J.; Rastegar, S.; Rindone, F.; Hartmann, V.; Stotzka, R.; García, A.; Wezel, J. v.; Mikut, R.; Strähle, U. & Nienhaus, G. U.
An Ensemble-Averaged, Cell Density-based Digital Model of Zebrafish Embryo Development Derived from Light-Sheet Microscopy Data with Single-Cell Resolution
Scientific Reports, 2015, 5, 1-10

